MA arsa - MA plot
Bir MA arsa bir uygulamasıdır Bland-Altman arsa görsel temsili için genomik verileri. Grafik, verileri M (log oranı) ve A ( ortalama ortalama ) ölçeklerine dönüştürerek ve ardından bu değerleri çizerek, iki örnekte alınan ölçümler arasındaki farkları görselleştirir . Başlangıçta iki kanallı DNA mikrodizi gen ekspresyon verisi bağlamında uygulanmış olsa da, MA grafikleri ayrıca yüksek verimli sıralama analizini görselleştirmek için de kullanılır .
Açıklama
Mikroarray verileri, boya birleştirme ve hibridizasyon verimliliklerindeki sistematik önyargıların yanı sıra DNA problarındaki ve diziyi tespit etmek için kullanılan baskı ucundaki diğer teknik önyargıları kontrol etmek için genellikle diziler içinde normalleştirilir. Bu sistematik varyasyonları en aza indirerek, gerçek biyolojik farklılıklar bulunabilir. Normalizasyonun gerekli olup olmadığını belirlemek için, Cy5 (R) yoğunluklarını Cy3 (G) yoğunluklarına göre çizebilir ve çizginin eğiminin yaklaşık 1 olup olmadığını görebiliriz. Temelde R vs'nin 45 derecelik bir G grafiği bir MA grafiğidir. MA grafiği, ortalama yoğunluk ('A') ile çizilen kırmızı / yeşil yoğunluk oranının ('M') dağılımının bir grafiğidir. M ve A aşağıdaki denklemlerle tanımlanır.
Bu nedenle M, yoğunluk oranının (veya log yoğunlukları arasındaki farkın) ikili logaritmasıdır ve A, grafikteki bir nokta için ortalama log yoğunluğudur. MA grafikleri daha sonra ham mikrodizi verilerinin yoğunluğa bağlı oranını görselleştirmek için kullanılır (mikrodiziler tipik olarak burada bir sapma gösterir, daha yüksek A daha yüksek | M | ile sonuçlanır, yani nokta ne kadar parlaksa numune ve kontrol arasında gözlenen fark o kadar muhtemeldir). MA arsa koyar değişken M üzerindeki y -Axis ve A üzerindeki x -Axis ve hızlı bir bakış sağlar dağıtım verilerinin.
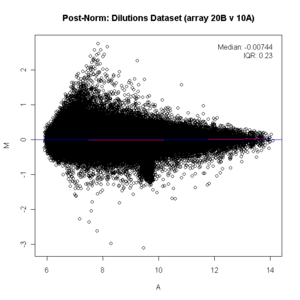
Pek çok mikrodizi gen ekspresyon deneyinde, genlerin çoğunun ekspresyonlarında herhangi bir değişiklik görmeyeceği varsayımı altında yatan varsayımdır; bu nedenle, log (1) 0 olduğundan, y ekseni ( M ) üzerindeki noktaların çoğunluğu 0'da bulunur. Durum böyle değilse, daha önce verilere LOESS gibi bir normalleştirme yöntemi uygulanmalıdır. istatistiksel analiz. (Aşağıdaki diyagramda, normalizasyondan önce sıfır işaretinin altındaki kırmızı çizgiye bakın, düz olmalıdır. Düz olmadığından veriler normalize edilmelidir. Normalleştirildikten sonra, kırmızı çizgi sıfır çizgisi üzerinde düzdür ve şu şekilde gösterilir: pembe siyah.)
Paketler
R yazılımı için çeşitli Bioconductor paketleri, MA grafikleri oluşturma olanağı sağlar. Bunlar affy (ma.plot, mva.pairs), limma (plotMA), marray (maPlot) ve edgeR (maPlot) içerir.
Caroline CRAN R paketindeki raPlot işlevi kullanılarak benzer "RA" grafikleri oluşturulabilir .
Genleri M, A ve p değerlerine göre filtrelemek, adlara göre veya bir kementle aramak ve seçilen genleri kaydetmek için etkileşimli bir MA grafiği, R-Parlak kodlu Geliştirilmiş MA Grafiği olarak mevcuttur .
R programlama dilinde örnek
library(affy)
if (require(affydata))
{
data(Dilution)
}
y <- (exprs(Dilution)[, c("20B", "10A")])
x11()
ma.plot( rowMeans(log2(y)), log2(y[, 1])-log2(y[, 2]), cex=1 )
title("Dilutions Dataset (array 20B v 10A)")
library(preprocessCore)
#do a quantile normalization
x <- normalize.quantiles(y)
x11()
ma.plot( rowMeans(log2(x)), log2(x[, 1])-log2(x[, 2]), cex=1 )
title("Post Norm: Dilutions Dataset (array 20B v 10A)")
Ayrıca bakınız
Referanslar
- ^ Robinson, MD; McCarthy, DJ; Smyth, GK (11 Kasım 2009). "edgeR: dijital gen ekspresyon verilerinin diferansiyel ifade analizi için bir Bioconductor paketi" . Biyoinformatik . 26 (1): 139–140. doi : 10.1093 / biyoinformatik / btp616 . PMC 2796818 . PMID 19910308 .
- ^ Sevgi, Michael I; Huber, Wolfgang; Anders, Simon (5 Aralık 2014). "DESeq2 ile RNA-seq verileri için kat değişiminin ve dağılımının ılımlı tahmini" . Genom Biyolojisi . 15 (12): 550. doi : 10.1186 / s13059-014-0550-8 . PMC 4302049 . PMID 25516281 .
- ^ YH Yang , S Dudoit , P Luu, DM Lin, V Peng, J Ngai, TP Speed . (2002). CDNA mikrodizi verileri için normalleştirme: tekli ve çoklu slayt sistematik varyasyonunu ele alan sağlam bir bileşik yöntem. Nucleic Acids Research cilt. 30 (4) s. E15.
- ^ Dudoit, S , Yang, YH , Callow, MJ, Hız, TP . (2002). Kopyalanmış cDNA mikrodizi deneylerinde farklı şekilde ifade edilen genlerin tanımlanması için istatistiksel yöntemler. Stat. Günah. 12: 1 111–139